2024年1月9日|癌研究
高精度シークエンシングにより、がんに関連するマイクロサテライト不安定性の検出が可能に
< Return to Japan blog | < Return to Main blog
がん生物学の複雑な状況の中で、マイクロサテライト不安定性(MSI)の研究は、診断方法や治療介入を改善するのに役立つ深い洞察を提供する可能性のある有望なフロンティアとして浮上してきました。ショートタンデムリピート(STR)の一種であるマイクロサテライトは、ヒトゲノム中に散在する小さな反復DNA配列であり、遺伝子発現や他の細胞プロセスの制御において極めて重要な役割を果たしています1。しかし、ゲノムの完全性を守る細胞のDNAミスマッチ修復(MMR)システムが機能不全に陥ると、細胞は分裂時にマイクロサテライトの長さを調節する能力を失い、その結果、MSIを発症します2。最近の大規模シークエンシング、特にがんゲノム解析の取り組みにより、大腸がん、子宮内膜がん、胃腺がんなど、さまざまなタイプの腫瘍におけるMSIの有病率が明らかになり、MSIの医学的意義が明らかになりつつあります2。
がん治療の指針となりうるマイクロサテライト不安定性の重要性
特に大腸癌や子宮内膜癌では、腫瘍中のMSIレベルの上昇は、免疫療法治療に対する患者の反応性の良い予測因子であることが示されています3,4。このような理由から、多様な種類の腫瘍におけるMSIの存在とその意味を研究することは、がん研究において衝撃的な進歩をもたらす絶好の機会となります。大腸がんや子宮内膜がんにおけるMSIステータスの臨床検査は、MMR関連タンパク質の免疫組織化学的検査か、少数の既知のマイクロサテライト遺伝子座のPCR検査に依存していますが、最近の研究では、NGS(次世代シーケンサー)に基づくアプローチが、より広範ながん種におけるMSIの検出に役立つ可能性があることが示されています5。
しかし、マイクロサテライト配列は本質的に反復的であるため、従来のシーケンス法ではMSIを正しく検出することができないという大きな課題があります。そのため、がん種を問わずMSIの状態を予測し、治療戦略に反映させる可能性のあるNGSの利用は、これらの反復領域を正確に読み取るシーケンス装置の能力に依存しています。
こうした課題に真正面から取り組むため、カナダのケベック州にあるマギル大学のDOvEEgene(Detecting Ovarian and Endometrial cancer Early using Genomics)プロジェクトの科学者チームは最近、極めて高精度(Q40+)なSBB(Sequencing by Binding)ケミストリーを備えた新しいPacBio Onsoショートリードシーケンスシステムの能力を調査し、SBS(Sequencing by Synthesis)に基づく標準的なアプローチよりもMSIをより効果的に検出できるかどうかを試みました。この取り組みから得られた予備的結果は、2023年10月にロンドンで開催されたthe Early Detection of Cancer conferenceで発表され、非常に有望視されています。
DOvEEgene 卵巣がんと子宮内膜がんの早期発見を目指して
DOvEEgeneプロジェクトは、卵巣癌と子宮内膜癌に焦点を当てています。早期発見が急務であることから、このプロジェクトは、これらのがんをできるだけ早期に発見するという野心的な目標を掲げて発足しました。マギル大学を拠点とするチームは、低コストで侵襲性が低いだけでなく、子宮頸がんの発見に革命をもたらしたパップテストのように、広く利用できる検査を開発することによって、これを達成したいと考えています。
MSI検出戦略の新たな前進としての高精度ショートリードシーケンス
DOvEEgeneプロジェクトの成功は、非常に高い感度と特異性の両方で癌変異を正確に検出できるかどうかにかかっています。これを達成するために、研究者らは、既存のショートリード・シーケンス技術と比較して、短いタンデムリピート領域でより優れた性能を達成できるPacBio Onsoシステムを活用しています。Onsoは、シーケンシングサイクルの質問ステップと組み入れステップを切り離すことで、分子傷跡を減らし、がんゲノムのこのような困難な反復領域でもポリメラーゼの性能を向上させます。
既存の技術に対するOnsoシステムの真の能力を調査するため、DOvEEgeneの科学者たちは、被験者のグループから唾液サンプルとともに子宮内ブラシサンプルを採取しました。その後、これらのサンプルからDNAを抽出し、卵巣がんや子宮内膜がんに関与することが知られている変異体をターゲットにしたパネルを用いて、ライブラリーの前処理とハイブリッドキャプチャーを行いました。ライブラリーはNovaSeq(S4フローセル)とPacBio Onsoシーケンスプラットフォームの両方でシーケンスされました。その後、481検体からなるデータセットでの広範なトレーニングとテストを通じて開発された機械学習アルゴリズムが採用され、ゲノムデータに基づいて、被験者ががんに罹患している確率を予測し、関連するMSIを検出しました。
見えるもの、見えないものに対する自信を深める
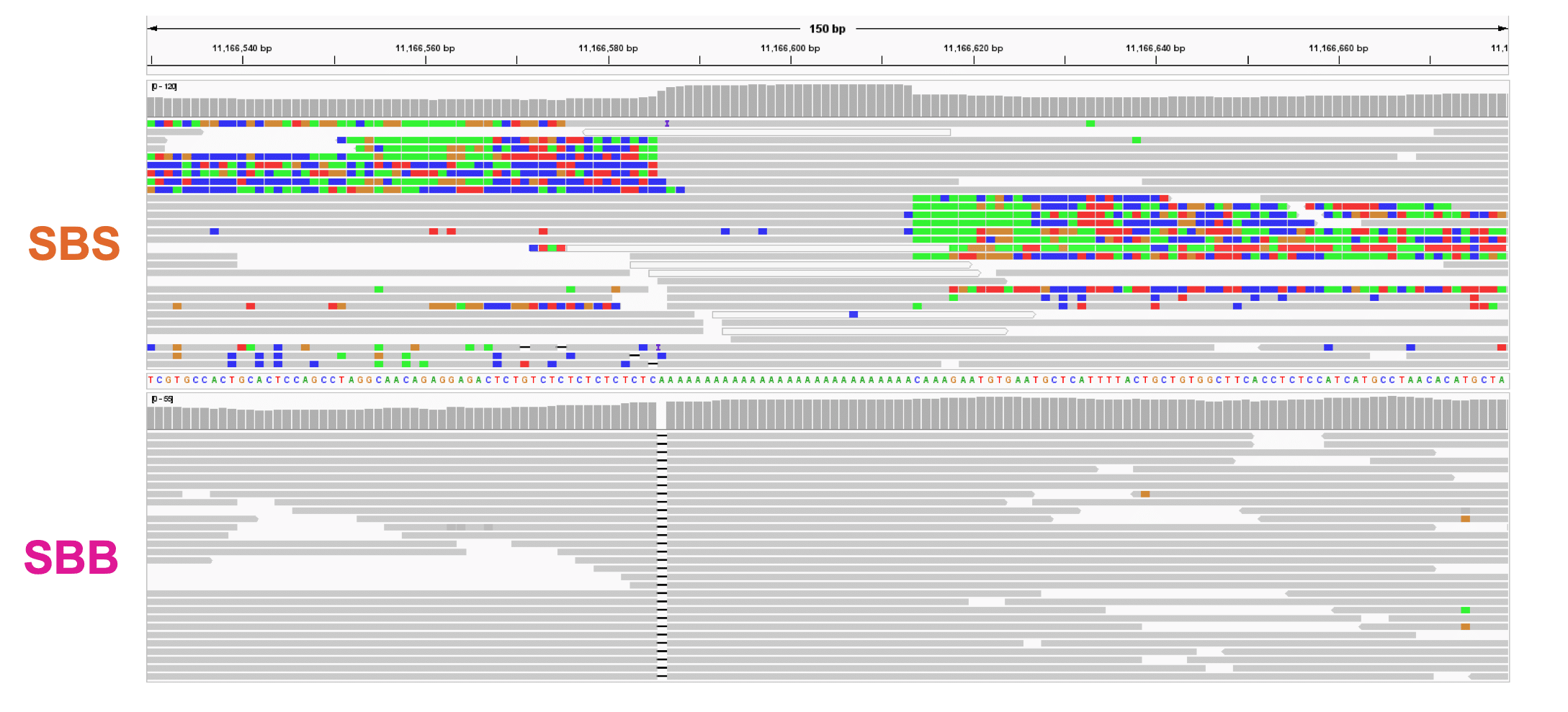
ホモポリマー、特にポリAリピートは、ゲノムの中で最も頻度が高く、変異が起こりやすいタイプのマイクロサテライトですが、MSI検出のための既存のシーケンス法では、これらの領域を正確に特徴付けることに長い間苦労してきました6。対照サンプルで以前に実証されたように、ホモポリマーを通して配列決定するOnsoシステムの強力な能力は、この制限を克服しています(HG002で実施した別の比較研究からの例を図1に示す)。また、ホモポリマーの長さをより正確に列挙できるため、MSIをより正確に検出する基礎となる位相差の問題も軽減されました。
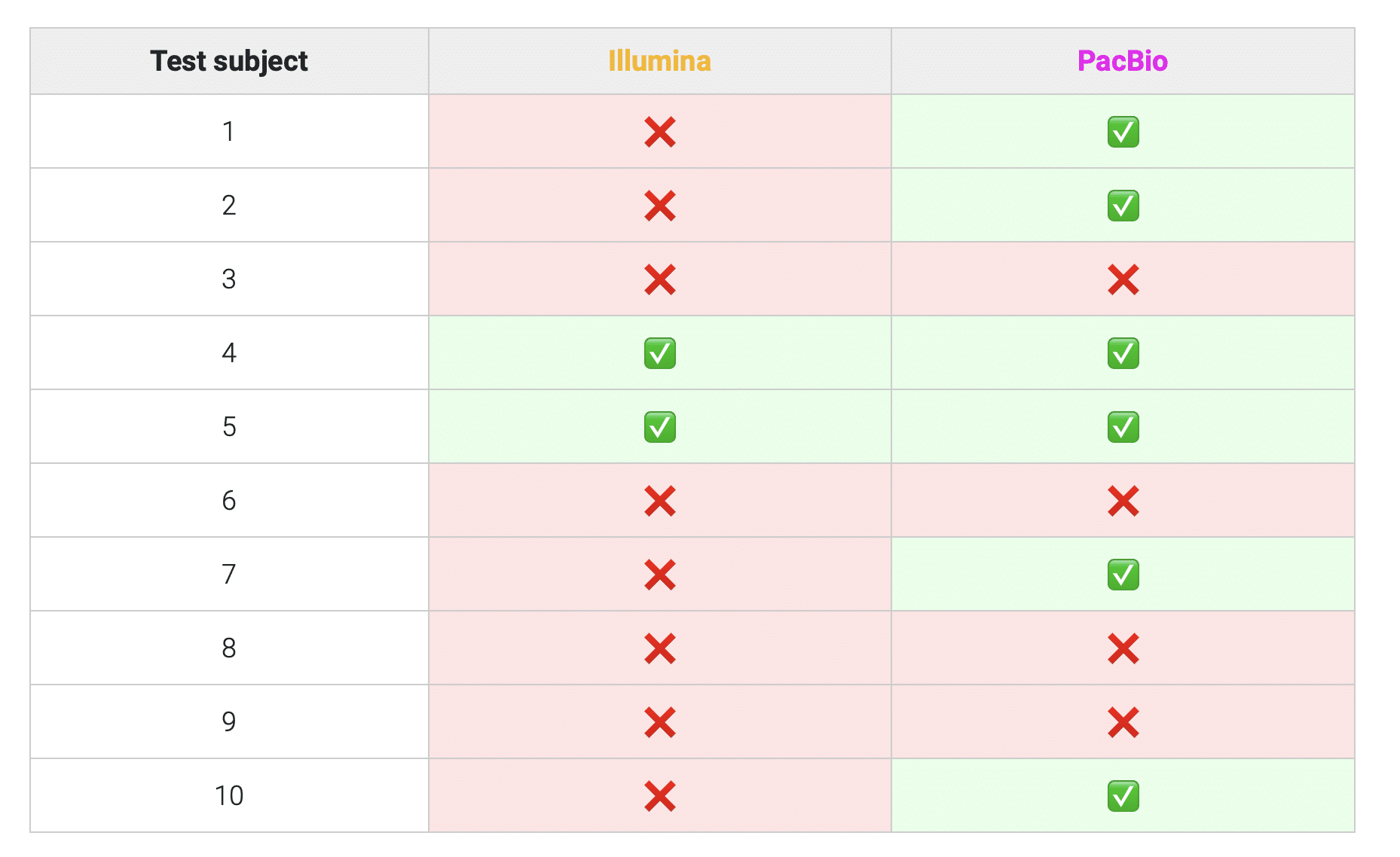
McGillチームの予備データによると、PacBio Onsoシステムは、既存のSBS法よりもDOvEEgene標的パネル領域の不安定なマイクロサテライト遺伝子座をより多く同定しており、これはこれらの反復領域におけるOnsoのシーケンス性能が向上した結果である可能性があります(表1)。OnsoによるMSI検出の増加の根拠を探り、標準的な臨床検査に対する性能をベンチマークするためには、さらなる研究が必要であるが、McGill大学の研究チームによるこれらの知見は、早期がん検出の研究におけるOnsoとその中核であるSBB化学の有望な将来を示唆するものであり、将来、ゲノムアッセイを臨床に適用する際の精度と信頼性を向上させる上で、シーケンシング技術が果たす重要な役割を強調しています。
オンソシステムとSBBに代表されるゲノミクスとシーケンス技術の絶え間ない進歩は、研究者が精密医療と個別化がん治療において新たな探求の道を開くことを可能にする可能性を秘めています。早期発見を可能にするための変革の道を歩むがん研究者コミュニティをサポートし続けますので、AACR2024での最新情報にご期待ください。
References
- Bagshaw ATM. Functional Mechanisms of Microsatellite DNA in Eukaryotic Genomes. Genome Biol Evol. 2017 Sep 1;9(9):2428-2443. doi: 10.1093/gbe/evx164. PMID: 28957459; PMCID: PMC5622345.
- Bonneville, R. et al.Landscape of Microsatellite Instability Across 39 Cancer Types. Precision Oncology. DOI: 10.1200/PO.17.00073.Published online October 3, 2017.
- Motta R, Cabezas-Camarero S, Torres-Mattos C, Riquelme A, Calle A, Figueroa A, Sotelo MJ. Immunotherapy in microsatellite instability metastatic colorectal cancer: Current status and future perspectives. J Clin Transl Res. 2021 Aug 4;7(4):511-522. PMID: 34541365; PMCID: PMC8445628.
- Green AK, Feinberg J, Makker V. A Review of Immune Checkpoint Blockade Therapy in Endometrial Cancer. Am Soc Clin Oncol Educ Book. 2020 Mar;40:1-7. doi: 10.1200/EDBK_280503. PMID: 32213091.
- Stephen J Salipante, Sheena M Scroggins, Heather L Hampel, Emily H Turner, Colin C Pritchard, Microsatellite Instability Detection by Next Generation Sequencing,Clinical Chemistry, Volume 60, Issue 9, 1 September 2014, Pages 1192–1199, https://doi.org/10.1373/clinchem.2014.223677
- Caitlin A. Loh, Danielle A. Shields, Adam Schwing, Gilad D. Evrony. High-fidelity, Large-scale Targeted Profiling of Microsatellites. bioRxiv 2023.11.28.569106; doi: https://doi.org/10.1101/2023.11.28.569106
PacBioに問い合わせる