AUGUST 8, 2023 | SEQUENCING 101
シーケンシング入門:SBBシーケンス
< Return to Japan blog | < Return to Main blog

10年以上前に次世代シーケンサー(NGS)が登場して以来、ショートリードのシーケンス精度はわずかな改善しか見られていません。ほとんどの用途で「十分」と考えられるレベルの精度を達成したため、NGS開発の多くはコストとスループットの最適化に集中してきました。その結果、重要な発見を可能にする能力とともに、ショートリードシーケンスの基本的な未処理リード精度(1,000塩基に1塩基、またはQ30)は、ほぼ頭打ちとなりました。
Onso short-read sequencing systemにPacBio sequencing by binding (SBB)テクノロジーを導入することで、10,000塩基に1塩基以下のエラー率(Q40+)の極めて正確なリードを生成することができます!
SBBは、研究者がゲノミクスで可能なことの限界を押し広げ続けるために、長い間必要とされてきたショートリードシーケンスにおける根本的なブレークスルーを意味します。 しかし、これはシーケンス全般にとって何を意味するのでしょうか? SBBシーケンスとは何か、どのように機能するのか、そしてその破壊的な能力がショートリードシーケンスによる研究の未来に何を意味するのか、私たちと一緒に考えてみましょう。
違いはケミストリーにある
近年のロングリードシーケンス技術の大きな進歩とは対照的に、既存のショートリードシーケンスでは、その核となるSBS(Sequencing by Synthesis)ケミストリーにほとんど変更が加えられていません。その結果、多くの研究イニシアチブと方法論は、この古い技術が基盤としている化学的性質によって生じる精度の制約によって制限されています。しかし、PacBio SBBテクノロジーの中核をなす画期的な新しいケミストリーは、こうした制約を完全に覆す力を持っています。
従来のSBSテクノロジーとは異なり、SBBシーケンスではシーケンスサイクルの各フェーズで最適化された条件を使用するため、生のリードエラーがほぼ完全に排除されます。一見するとわずかなことに見えますが、このケミストリーの決定的な違いは、私たち全員が待ち望んでいたショートリードシーケンスの精度におけるブレークスルーを構成しています。
SBBシーケンスとは何ですか?
PacBio SBBシーケンシングはQ40+のショートリードシーケンシングテクノロジーで、蛍光標識されたヌクレオチドがDNA鎖上のポリメラーゼに結合(ただし取り込まれない)したときの光シグナルを測定することで機能します。その後、コンピューターがこれらの光信号を対応する塩基のベースコールと照合する。他のショートリード技術とは異なり、SBBはシークエンシングプロセスの結合ステップとその後の伸長ステップを分離することで、分子アーチファクトによるエラーを排除しています。
SBBシーケンシングの仕組み
次世代SBBシーケンシングケミストリーは、4つの主要なステップ(開始、検出、活性化、取り込み)によって進行します。
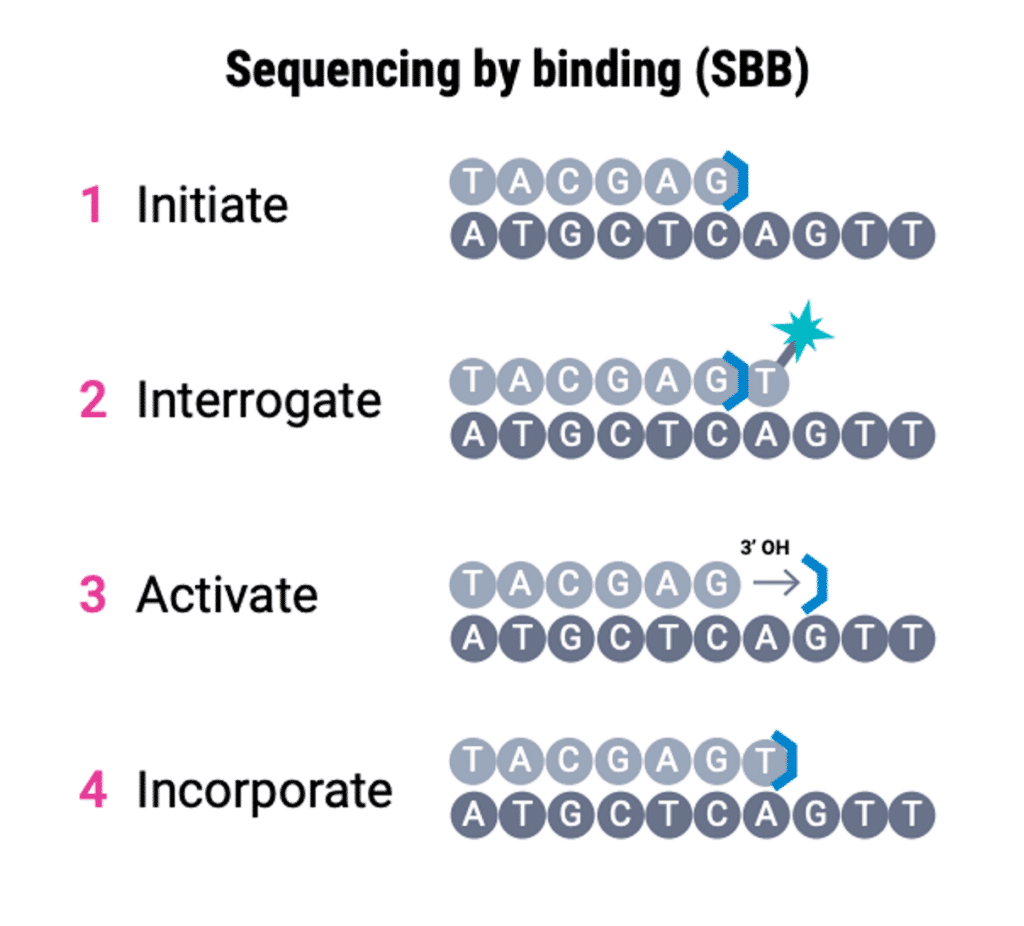
SBB技術では、DNA鎖上のポリメラーゼによってシーケンスが開始され、3’末端に可逆的なブロッカーが付加され、塩基が成長中の鎖に完全に組み込まれるのを防ぐ(ステップ1)。その後、蛍光タグが付けられた塩基がフローセルに流れ込みます。従来のSBSシーケンスとは異なり、これらのタグ付き塩基はブロッカーを含みません。適切なタグ塩基がDNA鎖に結合すると、蛍光シグナルが発せられ、強力な光学系を用いてこれを検出します(ステップ2)。SBSシーケンシングとの決定的な違いとして、このタグ塩基はDNA鎖に取り込まれるのではなく、塩基シグナルがシーケンシング装置によって捕捉された後に洗い流されます。その後、可逆的ブロッカーの除去によってヌクレオチドの3’末端が活性化されます(ステップ3)。これにより、次のサイクルの適切なタイミングまで、追加塩基の取り込みを防ぎ、非標識でブロッカーの付加されたヌクレオチドを確実に取り込むことができます(ステップ4)。
SBBと従来のSBSとの違いは?
分子に傷がつかない
SBBシーケンスの画期的な精度の背後にある重要な技術革新は、ベースコールシグナルを発する蛍光タグ付きヌクレオチドが、一般的なSBSシーケンスでのようにDNA鎖に直接組み込まれないという事実にあります。これらのステップを分離することで、2つのステップを別々に最適化することが可能になり、ポリメラーゼの忠実度、シグナル対ノイズレベルが向上し、蛍光タグが伸長中のDNA鎖から除去される際に従来のSBSシーケンシングで発生する分子の傷の有害な影響を排除することができます。SBSシーケンスでは、各蛍光タグを除去するために必要な切断により、各ヌクレオチド塩基に付着したリンカーアームが残ります(分子の傷と呼ばれます)。これらの分子残基は、何サイクルにもわたって、DNA鎖を読み取ろうとするポリメラーゼを妨害し、読み取りの精度に悪影響を及ぼします。
最小限の重複
PacBio SBBシーケンスは、分子の傷によるシーケンスエラーの蓄積を回避するだけでなく、従来のショートリード技術に比べて重複率が非常に低くなっています。重複するリードとは異なり、重複配列は解析に付加的な情報価値をもたらさないため、重複排除と呼ばれるプロセスでバイオインフォマティクスによって除去されるのが一般的です。SBBシーケンスではこのような重複配列の数が少ないため、重複排除プロセスに必要な総データ量と計算リソースが少なくなり、全体としてより合理的な解析が可能になります。
最小限のインデックスホッピング
SBBシーケンスでは、マルチプレックスサンプルにおけるインデックスのホッピングは最小限に抑えられます。SBBとは異なり、従来のSBSシーケンスで使用されるパターン化フローセルの増幅法では、シーケンス中にユニークインデックスの一部がサンプルプール間でクロスオーバーする可能性があります。その結果、一方のサンプルからのリードが他方のサンプルに誤って割り当てられ、データのノイズが増加します。PacBio SBBシーケンスにはこの問題はありません。
次世代レベルの精度
分子の傷をなくし、重複を減らし、インデックスのホッピングを最小限に抑えることで、SBBシーケンスは、10,000塩基に1塩基以下(Q40+)という前例のないエラー率で、驚異的なリード精度を実現します。
Q40+の精度は、ショートリードシーケンスの将来にとって何を意味するのか?
市場の他のベンチトップシーケンサー†と比較して最大15倍の精度を持つSBBケミストリーとOnsoシステムは、ショートリードシーケンス技術で可能なことに革命を起こしています。
しかし、このような画期的な精度はどのように測定され、それによって何が可能になるのでしょうか?
シーケンスリードの精度は、ゲノム研究者がPhred quality score(略してQスコア)と呼ぶものを用いて測定されます。Qスコアは、シーケンサーが誤った塩基判定を行う可能性に基づいています。例えば、Q10の品質スコアは10回に1回のエラー率を表し、90%の精度に相当する。Q20のスコアは100分の1のエラー率で99%の精度を表します。この指標は対数スケールで表されるため、この2つの品質スコアの間を移動すると、シーケンス精度が10倍に跳ね上がることになります。
PacBio OnsoシステムでのSBBシーケンスでは、≥Q40で90%の塩基が返されます。これは、従来のSBSシーケンステクノロジーで達成されていたQ30スコアから飛躍的に向上しています。Q30からQ40の精度への移行は、1,000塩基に1塩基のエラー率から10,000塩基以上に1塩基のエラー率へのジャンプを意味します!多くの点で、精度が1桁上がれば、発見も1桁増える可能性があります。
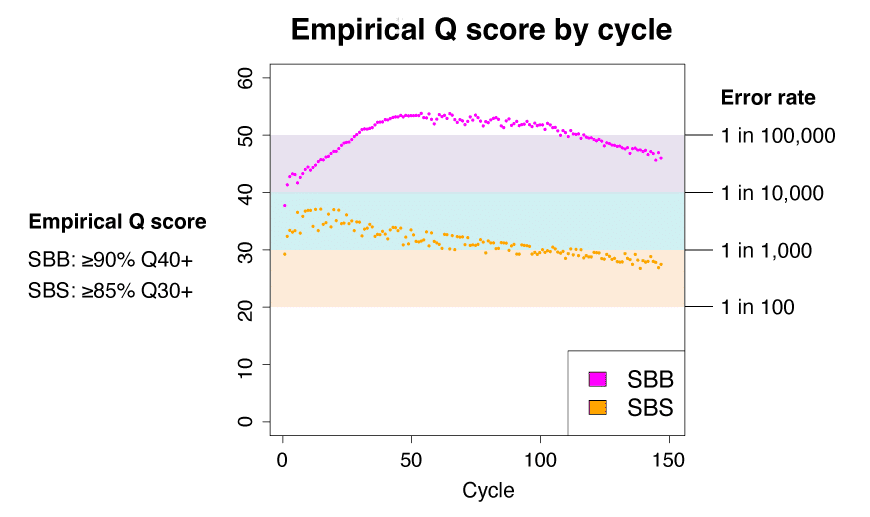
この驚くべきQ40+レベルの精度により、Onsoユーザーはより自信を持ってゲノムのシーケンスを行うことができ、リキッドバイオプシー、遺伝子編集、希少な体細胞バリアント、感染症における低レベルの耐性変異など、干し草の山から針を見つけようとするあらゆる研究アプリケーションにおいて大きな発見の扉を開くことができます。OnsoシステムのSBBが持つ並外れた感度は、従来のショートリードシーケンス法では見逃されていた極めて希少かつ重要なバリアントの検出限界を劇的に引き下げます。
例えば、SBBシーケンスのパワーにより、研究者は従来のSBSシーケンシングで必要とされるシーケンシングの最大4倍少ないシーケンシングを、同等以上の感度を維持しながら行うことができます。また、SBBシーケンシングでは、性能に影響を与えることなく、7倍低い濃度のサンプルをフローセルに流すことができるため、研究者は将来の研究のために貴重なサンプルをより多く保存することができます††。
精度の時代へようこそ
SBBケミストリーの驚くべき利点は、ショートリードシーケンス技術に期待できる精度と感度のレベルのパラダイムシフトを意味します。SBBシーケンシングによるQ40+の精度の価値は、個人、集団、グローバルヘルスのアプリケーションにおいて、計り知れない発見の可能性を秘めています。 SBBにより、精度の時代が到来しました。
SBB + Onsoを試す準備はできましたか?
†CHM13 Sequencing. Empirical Q scores evaluated on 99% of bases for each cycle.
††Compared to NextSeq 1/2k and NovaSeq 6000 S1 input requirements with initial library pool concentration of 500-5000 pM. Assumes library insert size of 500 bp for mass calculation.
PacBioに問い合わせる